'20/01/12更新:コードを見易くするため、グラフ化の箇所は関数化して分けた。
本記事では、Pythonの多目的最適化のフレームワークのひとつ「Platypus(カモノハシ)」を利用して、その中に含まれるGAアルゴリズムのひとつ「NSGAII(Non dominated Sorting Genetic Algorithms II)」の使用例である。
使用した分析データは、sklearnに含まれている「生理学的特徴と運動能力の関係」である。Python scikit-learn付属の多目的用のデータ「Linnerud dataset」をcsvファイル化する - HK29’s blog
目的関数3つの回帰モデル(回帰式)を各々作成する。作成例は右記リンク先を参照→Python scikit-learnによる交互作用の項を考慮した重回帰分析 - HK29’s blog
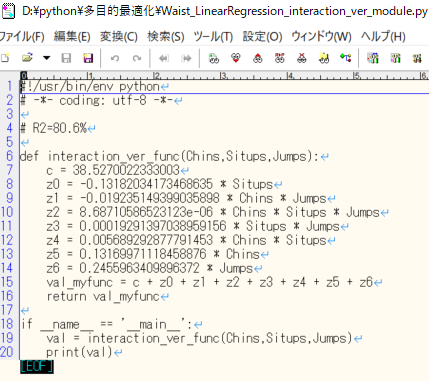
その結果、下図のような線形重回帰分析の回帰式を自作モジュールとして作成する。
本記事のコードを実行すると下図の3つのファイルを出力する。
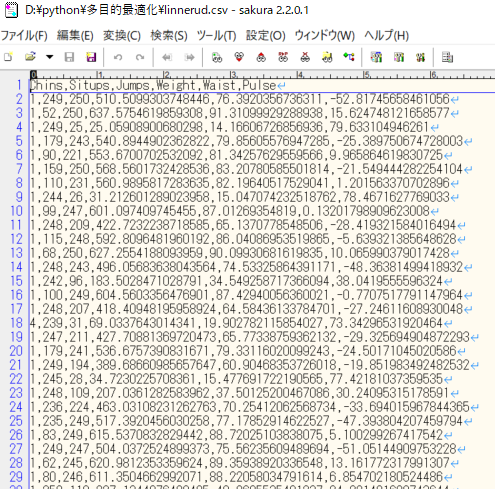
1. 各世代で得られた目的値を説明変数と共にcsvファイルに出力する
ねらいの目的値に対して、得られた設計値を知ることが出来る。
2. 複数の目的関数を横軸,縦軸にしてパレート解として可視化
下図は使用したデータが例としてよくなかったが、本来、トレードオフの関係にあるデータを分析すればパレート解まで確認できる。目的関数が3つ以上の場合で、それぞれ可視化したい場合はそれ用に編集する。
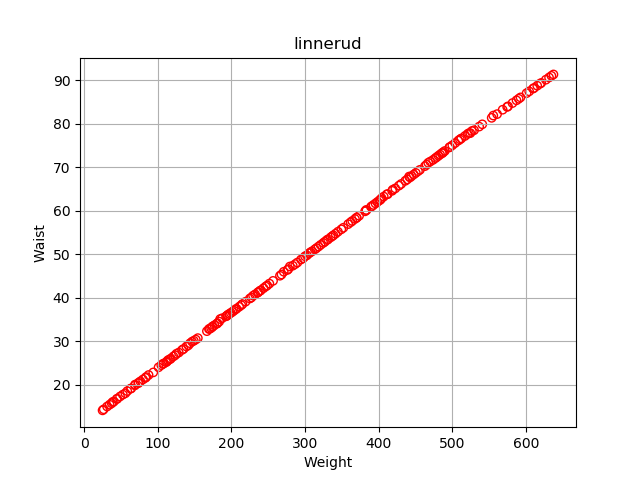
3. 全データを散布図にして可視化
一応、上記2がなくても下図で全ての相関関係を視覚的に確認できる。

■本プログラム
#!/usr/bin/env python # -*- coding: utf-8 -*- # https://platypus.readthedocs.io/en/latest/getting-started.html#defining-unconstrained-problems import sys sys.path.append(".") import Weight_LinearRegression_interaction_ver_module as myfunc1 # 体重の回帰式 import Waist_LinearRegression_interaction_ver_module as myfunc2 # 腹囲の回帰式 import Pulse_LinearRegression_interaction_ver_module as myfunc3 # 心拍数の回帰式 from platypus import NSGAII, Problem, Real, Integer import pandas as pd import matplotlib matplotlib.use('Agg') import matplotlib.pyplot as plt import seaborn as sns # 目的関数を作成して返す関数:自作モジュールを読み出す場合 def my_function_from_module(vars): Chins = vars[0] # x1 懸垂の回数 Situps = vars[1] # x2 上体反らしの回数 Jumps = vars[2] # x3 ジャンプ objective_function1 = myfunc1.interaction_ver_func(Chins, Situps, Jumps) objective_function2 = myfunc2.interaction_ver_func(Chins, Situps, Jumps) objective_function3 = myfunc3.interaction_ver_func(Chins, Situps, Jumps) return [objective_function1, objective_function2, objective_function3] # パレート解を図示する関数 def plot_scatter(algorithm, out_file_name, my_color): my_file_name = out_file_name + '_NSGAII' x_list = [s.objectives[0] for s in algorithm.result] y_list = [s.objectives[1] for s in algorithm.result] fig = plt.figure(figsize=(8,8)) #dpi=120) ax = fig.add_subplot(1,1,1) ax.set_title('Multi-objective optimization', fontsize=14) #plt.xlabel("$f_1(x)$") #plt.ylabel("$f_2(x)$") ax.set_xlabel(my_xlabel, fontsize=16) ax.set_ylabel(my_ylabel, fontsize=16) if my_xrange: ax.set_xlim([my_xrange[0], my_xrange[1]]) if my_yrange: ax.set_ylim([my_yrange[0], my_yrange[1]]) ax.scatter(x_list, y_list, facecolors='none', edgecolors=my_color, label="NSGAII" + '\n Pareto frontier') ax.legend(loc='best', fontsize=14) ax.grid(which='both') ax.tick_params(labelsize=14) plt.tight_layout() plt.savefig(my_file_name + '_Pareto_frontier.png') plt.close() # 行列散布図を作成する関数 def plot_matrix_scatter(label, DF, my_sequential_colormap): print("plot_matrix_scatter ...") sns.set(style="ticks", font_scale=1.2, palette=my_sequential_colormap, color_codes=True) g = sns.pairplot(DF, diag_kind="hist") g.fig.suptitle(label) g.fig.subplots_adjust(top=0.9) plt.grid(True) plt.savefig(label + "_Matrix_scatter.png") plt.close() # 最適化の過程をcsvファイルに出力する関数(この中で行列散布図の作成する関数も呼び出す) def create_Summary_data(Chins, Situps, Jumps, algorithm, my_sequential_colormap): my_file_name = out_file_name + '_NSGAII' column_name_list = ['Chins', 'Situps', 'Jumps', 'Weight', 'Waist', 'Pulse'] column_name_list = ','.join(column_name_list) row_list=[] with open(my_file_name + '.csv', 'w') as f: f.writelines(column_name_list) f.write('\n') for i in range(len(algorithm.result)): row_list.append(Chins.decode(algorithm.result[i].variables[0])) row_list.append(Situps.decode(algorithm.result[i].variables[1])) row_list.append(Jumps.decode(algorithm.result[i].variables[2])) row_list.extend(algorithm.result[i].objectives[:]) print('NSGAII epoch' +str(i+1) + ' -> ' + str(row_list)) row_list_str = ','.join(map(str, row_list)) f.writelines(row_list_str) f.write('\n') row_list=[] df = pd.read_csv(my_file_name + '.csv') plot_matrix_scatter(my_file_name, df, my_sequential_colormap) # メイン関数:platypusフレームワークによる多目的最適化 def main(my_color, my_sequential_colormap): problem = Problem(3, 3) # define 3 inputs and 3 objective (and no constraints) problem.directions[:] = [Problem.MAXIMIZE, Problem.MINIMIZE, Problem.MINIMIZE] # Problem.MINIMIZE Chins = Integer(1, 17) # x1 Situps = Integer(50, 250) # x2 Jumps = Integer(25, 250) # x3 problem.types[:] = [Chins, Situps, Jumps] problem.function = my_function_from_module print(problem.function) algorithm = NSGAII(problem, 200) # 200 is the population size algorithm.run(10000) # evaluation number print(algorithm) plot_scatter(algorithm, out_file_name, my_color) create_Summary_data(Chins, Situps, Jumps, algorithm, my_sequential_colormap) # パラメータの設定 if __name__ == '__main__': # parameter out_file_name = 'linnerud' my_xlabel = 'xlabel' my_xscale = 'linear' my_xrange = () # グラフのレンジを設定。空タプル()でAuto my_ylabel = 'ylabel' my_yscale = 'linear' my_yrange = (0, 100) # グラフのレンジを設定。(最小値, 最大値) my_colors = ["r", "g", "b", "c", "m", "y", "k", "w"] my_sequential_colormaps = ["spring", "summer", "autumn", "winter", 'bone', "copper", "plasma", "magma", "cividis", "hsv"] # call function main(my_colors[0], my_sequential_colormaps[0]) print('finished')
●参考リンク
●関連リンク
以上
<広告>
リンク