本記事ではオープンソースのライブラリ「Optuna」を使用した雛形コードを載せました。インストールは、次のようにcondaで行えます。
conda install -c conda-forge optuna
分析内容は、ボストンデータセットの住宅価格を目的関数に、scikit-learnのニューラルネットワーク回帰分析のハイパーパラメータの調整です。ハイパーパラメータとは、活性化関数や隠れ層の数、ニューロン数などの手法のパラメータのことです。これらは一意に決まるものではなく、分析するデータに依ります。それを自動で調整するのが「Optuna」です。本雛形コードでは、活性化関数、隠れ層、ニューロン数、ソルバーの4つとしました。
解析結果の例を以降に示します。最後に本雛形コードを記載しています。
下図中の「NN.py」が本プログラムで、それ以外は出力結果をファイルに保存します。
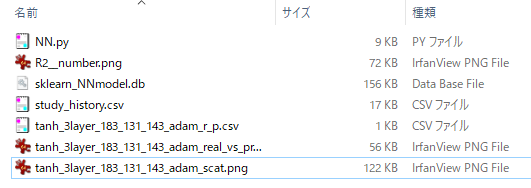
下図は「study_history.csv」の中身で、最適化過程をcsvファイルで出力します。
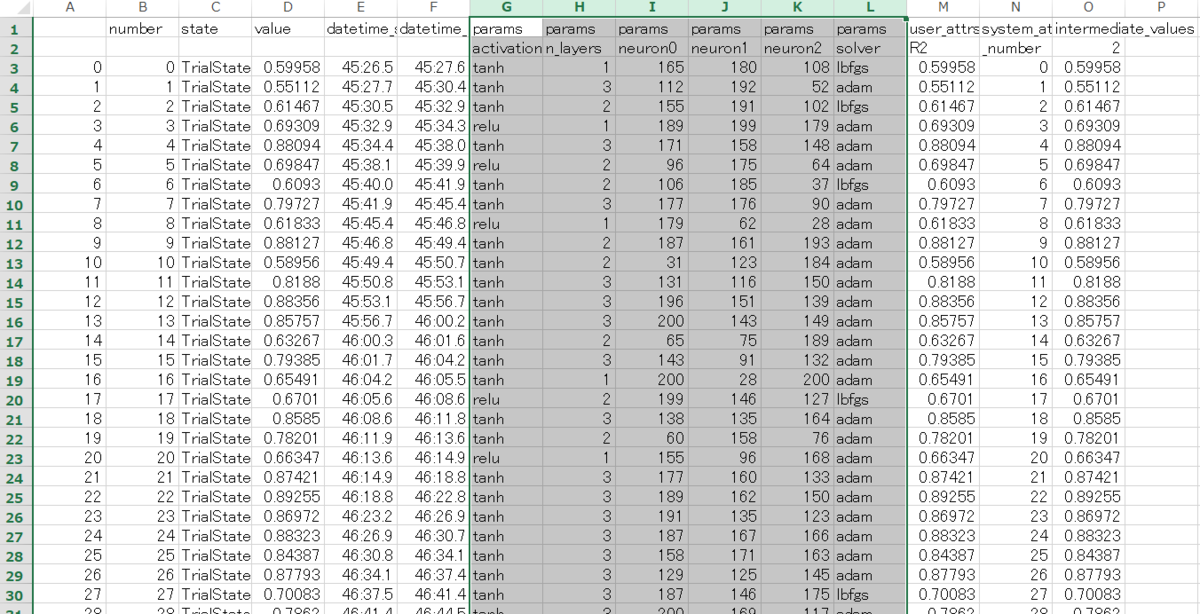
下図のように、縦軸の決定係数R2が1に近づいている様子がわかります。
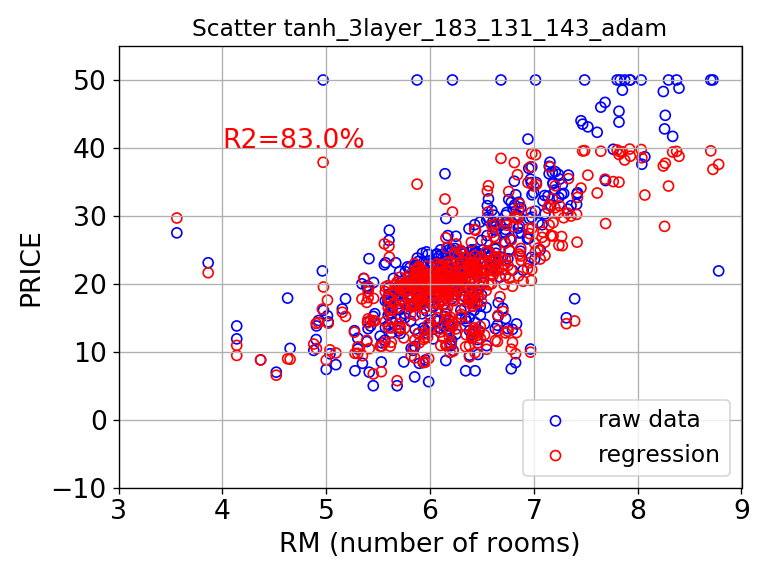
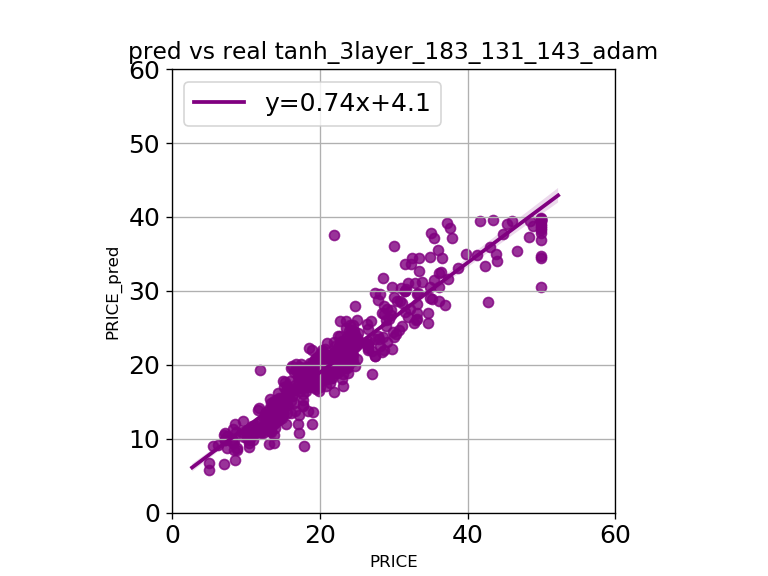
結果、ベターな回帰モデルが得られます。下図は、それを可視化したグラフで青色は生データ、赤色はR2を指標に選定されたハイパーパラメータによって実行したNNによる回帰モデルによる予測結果です。
下図は横軸が生値、縦軸が予測値です。一次近似式の傾きが1に近い程、傾向を再現しています。しかし、切片が0より遠いと傾向の感度のずれが大きいことを意味します。
各評価指標を下図のようにcsvで保存します。

そして、取得したハイパーパラメータはSQLiteの拡張子「.db」ファイルに記録できます。それを元に途中から再開することもできます。
■本プログラム
Anaconda環境であれば、コピペで実行できるはずです。参考までに、「Optuna」には学習過程で学習が進まなくなった場合に途中で停止する枝刈り機能があります。この発動判定にMedianPrunerの引数「n_startup_trials」と「n_warmup_steps」で調整できます。いくつか動作確認したところ、値を小さくすることで枝切り発生率は高くなります。しかし、最適解に到達しにくい傾向であり、この機能自体がハイパーパラメータになってしまう。今回のデータ分析では5くらいが目安でした。
#!/usr/bin/env python3 # -*- coding: utf-8 -*- # ver 20/01/03 import optuna import datetime, time import numpy as np import pandas as pd import scipy as sp import seaborn as sns import matplotlib matplotlib.use('Agg') import matplotlib.pyplot as plt import matplotlib.ticker as tick from sklearn import datasets from sklearn import neural_network from sklearn.metrics import mean_squared_error, explained_variance_score from sklearn.model_selection import cross_val_predict now = datetime.datetime.now() now = now.strftime("%y%m%d_%H%M%S") boston = datasets.load_boston() x = boston.data y = boston.target x_df = pd.DataFrame(x, columns=boston.feature_names) y_s = pd.Series(y) target_Yname = "PRICE" Xname = "RM" def create_model_NN(activation, n_layers, n_neurons, solver): hidden_layer_sizes=[] for i in range(n_layers): hidden_layer_sizes.append(n_neurons[i]) print('hidden_layer_sizes -> ' + str(hidden_layer_sizes)) model = neural_network.MLPRegressor(activation = activation, hidden_layer_sizes=hidden_layer_sizes, solver = solver) return model def objective(trial): # set param n_layer_max = 3 activation = trial.suggest_categorical('activation', ['tanh','relu']) n_layers = trial.suggest_int("n_layers", 1, n_layer_max) n_neurons=[] for i in range(n_layer_max): n_neurons.append(trial.suggest_int('neuron' + str(i), 20, 200)) solver = trial.suggest_categorical('solver', ['lbfgs','adam']) # create model model = create_model_NN(activation, n_layers, n_neurons, solver) # predict pred = model.fit(x,y) # score R2 = pred.score(x,y) trial.report(R2, i) trial.set_user_attr('R2', R2) if trial.should_prune(): raise optuna.structs.TrialPruned() return R2 def run_regr(method_name, regr): start = time.time() pred = regr.fit(x,y) R2 = pred.score(x,y) yp = regr.predict(x) rmse = np.sqrt(mean_squared_error(y,yp)) yp_cv = cross_val_predict(pred, x, y, cv=10) rmsecv=np.sqrt(mean_squared_error(y,yp_cv)) print('Method: {}'.format(method_name)) print('RMSE on the data: {:4f}'.format(rmse)) print('RMSE on 10-fold CV: {:.4f}'.format(rmsecv)) print(R2) print('R2:{:.1%}'.format(R2)) run_time = time.time() - start print('Run_Time: {:.1f}'.format(run_time)) yp_s = pd.Series(yp) plot_reg(x_df, target_Yname, y_s, yp_s, R2, run_time, title=method_name) plot_scatter(x_df, target_Yname, y_s, yp_s, R2, title=method_name) def plot_reg(x_df, target_Yname, y_s, yp_s, R2, run_time, title="original"): DF = pd.concat([y_s, yp_s], axis=1) DF.columns = [target_Yname, target_Yname + '_pred'] slope, intercept, r_value, p_value, std_err = sp.stats.linregress(y_s, yp_s) r_pearsonr, p_pearsonr = sp.stats.pearsonr(y_s, yp_s) ax = plt.figure(num=0, dpi=120).gca() plt.rcParams["axes.labelsize"] = 15 ax.set_title("pred vs real " + title, fontsize=14) sns.regplot(x = target_Yname, y = target_Yname + "_pred", data = DF, color="purple", line_kws={'label':"y={0:.2f}x+{1:.1f}".format(slope,intercept)}) ax.set_xlim(0, 60) ax.set_ylim(0, 60) ax.set_aspect('equal', adjustable='box') plt.grid(True) plt.legend(loc='upper left', fontsize=15) plt.tick_params(labelsize=15) plt.savefig(title + '_real_vs_pred.png') plt.close() buf1_str = ("slope, intercept, r_value, p_value, std_err, r_pearsonr, p_pearsonr, R2, run_time") buf2_list = [slope, intercept, r_value, p_value, std_err, r_pearsonr, p_pearsonr, R2, run_time] buf2_list_str = [str(n) for n in buf2_list] buf2_list_str_with_csv = ','.join(buf2_list_str) with open(title + "_r_p.csv", 'a') as f: f.write(buf1_str + '\n') f.writelines(buf2_list_str_with_csv) def plot_scatter(x_df, target_Yname, y_s, yp_s, R2, title="original"): fig = plt.figure(dpi=120) ax = fig.add_subplot(1,1,1) ax.set_title('Scatter ' + title, fontsize=14) ax.set_xlabel('RM (number of rooms)', fontsize=16) ax.set_ylabel(target_Yname, fontsize=16) ax.set_xlim([3, 9]) ax.set_ylim([-10, 55]) ax.scatter(x_df.loc[:,Xname], y, facecolors='none', edgecolors='b', label='raw data') ax.scatter(x_df.loc[:,Xname], yp_s, facecolors='none', edgecolors='r', label='regression') ax.legend(loc='lower right', fontsize=14) ax.tick_params(labelsize=16) plt.text(4, 40, 'R2={:.1%}'.format(R2), size = 16, color = "red") plt.grid(True) plt.tight_layout() #plt.show() plt.savefig(title + '_scat.png') plt.cla() # Clear axis plt.clf() # Clear figure plt.close() # Close a figure window def plot_R2graph(read_file, myheader, mycolumn, N): x_name, y_name = mycolumn df = pd.read_csv(read_file, header=myheader) #, skiprow=2) df_x = df[x_name] df_y = df[y_name] plt.figure(num=0, figsize=(12,8)) plt.title('the evolution of the coefficient of determination') plt.scatter(df_x, df_y, c="red") plt.plot(df_x, df_y, color="red") plt.xlabel('Number of Cycle') plt.ylabel('R^2') #for i, j in zip(df_x, df_y): # plt.text(i, j, '%.2f' % j, ha='center', va='bottom', color='b') plt.xlim([0, N]) plt.ylim([0, 1]) plt.gca().xaxis.set_major_locator(tick.MultipleLocator(1)) plt.grid(which='both') #plt.tick_params(labelsize=22) plt.tight_layout() #plt.show() plt.savefig(y_name + "_" + x_name + '.png') plt.close() if __name__ == '__main__': ### optuna study_name = 'test' myreset = True if myreset: # 真の場合:新規に実行する。 study = optuna.create_study(pruner=optuna.pruners.MedianPruner(n_startup_trials=5, n_warmup_steps=5), study_name = study_name, direction = 'maximize', storage = 'sqlite:///sklearn_NNmodel.db') else: # 偽の場合:もし、すでに前回の回帰モデルのファイル「.db」が存在すればそれを実行する。 study = optuna.create_study(study_name = study_name, direction = 'maximize', storage = 'sqlite:///sklearn_NNmodel.db', load_if_exists = True) study.optimize(objective, n_trials = 100) #study.optimize(objective, timeout=120) outfile = "study_history.csv" study.trials_dataframe().to_csv(outfile) pruned_trials = [t for t in study.trials if t.state == optuna.structs.TrialState.PRUNED] complete_trials = [t for t in study.trials if t.state == optuna.structs.TrialState.COMPLETE] print('Study statistics: ') print(' Number of finished trials: ', len(study.trials)) print(' Number of pruned trials: ', len(pruned_trials)) print(' Number of complete trials: ', len(complete_trials)) print('Best trial:') trial = study.best_trial print(' Value: ', trial.value) print(' Params: ') for key, value in trial.params.items(): print(' {0}: {1}'.format(key, value)) ### param after optimization print(study.best_params) print(study.best_value) print(str(study.best_params["n_layers"])) hidden_layers_list = [] neuron_name = '' for i in range(study.best_params["n_layers"]): hidden_layers_list.append(study.best_params["neuron"+str(i)]) neuron_name += '_' + str(study.best_params["neuron"+str(i)]) hidden_layers_tuple = tuple(hidden_layers_list) print(hidden_layers_tuple) ### run after optimization scatter_filename = study.best_params["activation"] + '_' \ + str(study.best_params["n_layers"]) + 'layer' \ + neuron_name + '_' + study.best_params["solver"] run_regr(scatter_filename, neural_network.MLPRegressor(activation = study.best_params["activation"], hidden_layer_sizes = hidden_layers_tuple, solver = study.best_params["solver"]) ) ### plot graph myheader = 1 mycolumn = ["_number", "R2"] plot_R2graph(outfile, myheader, mycolumn, len(study.trials))
(参考)
ModuleNotFoundError: No module named 'sqlalchemy' エラーが生じた場合
下記コマンドでインストールする。
pip install Flask-SQLAlchemy
以上
<広告>