クラスタリング、クラスター分析と言えば、k-meansが有名です。しかし、予めクラス(分類)数を指定する必要があります。この対策として、分類数を自動で決定するアルゴリズムはいくつか存在していて、「x-means」,「g-means」,「Star Clustering」などがあります。
本記事では、「x-means」と「g-means」の雛形コードと実行結果の比較例を載せました。この2つは、pyclusteringをインストールすることで使用できます。
■インストール方法
pipの場合
pip3 install pyclustering
Anaconda環境下の場合
conda install -c conda-forge pyclustering
■分析に使用した例題データ
scikit-learnに同梱されているワインデータセットを用いました。本来は、機械学習の分類用に用意されたデータセットで、説明変数(特徴量)が13項目あって、予め3つに分類された「target」があります。詳細は次のリンク先を参照sklearn.datasets.load_wine — scikit-learn 0.24.2 documentation
本記事では、分類ではなくてクラスター分析のため、教師データは使わずに分類するため、上記「target」を用いた分析はしません。(分析後の比較だけに使います)
■クラスタリングの実施例
先に、実施例について説明します。分析時に使用した説明変数の数が2, 3, 13項目の3つ場合について順番に説明します。
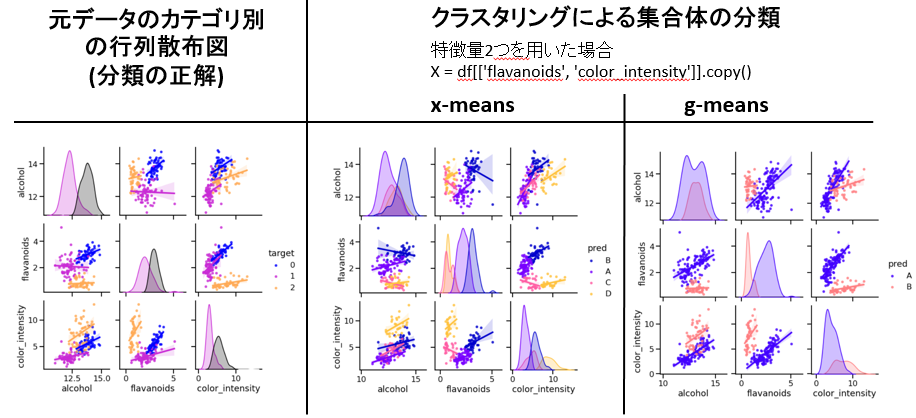
下図は、1つ目の例です。クラスター分析に特徴量2項目だけを用いた場合です。
下図左は元データで3つのカテゴリに分かれています(3つのクラス分類)。一方、下図中央のx-meansでの分析結果は4つにクラス分類され、下図右のg-meansの場合では2つにクラス分類されました。分類数が異なる結果になりました。しかし、それら集合体の分布は違和感はなく妥当に仕分けされたようにみえます。本来は正解データはわからないため、技術者本人が用途に応じて判断することになります。
下図は、2つ目の例です。クラスター分析に特徴量「3項目」を用いた場合です。
x-meansは3つにクラス分類され、g-meansは4つにクラス分類された結果です。

下図は、3つ目の例です。クラスター分析に特徴量全て13項目を用いた場合です。
x-meansは3つにクラス分類され、g-meansは7つにクラス分類された結果です。

上記3つの例は、クラスタリングに使用する説明変数の数を変更して検証しました。実は、本コードでは前処理としてデータを標準化しています。この前処理の有無でも結果は変わります。他には、クラスター分析したい説明変数間に桁が著しくことなるデータ群、例えば、対数データ群や何桁も小さい小数データ群などがある場合は標準化した方が吉です。また、実務においては外れ値が存在する場合も頭に入れておき、場合によってはそれらを削除する判断が必要なこともありえます。
■本プログラム
本コードはx-meansの場合の例です。g-meansを使用したい場合は、xmeansの箇所をgmeansに置換するだけでOKです。
from sklearn import datasets, preprocessing
import pandas as pd
from pyclustering.cluster.xmeans import xmeans
from pyclustering.cluster.center_initializer import kmeans_plusplus_initializer
import matplotlib.pyplot as plt
wine_data = datasets.load_wine()
df = pd.DataFrame(wine_data.data, columns=wine_data.feature_names)
df
X = df[['alcohol', 'flavanoids', 'color_intensity']].copy()
scaler = preprocessing.StandardScaler()
scaler.fit(X)
scaled_X = scaler.transform(X)
scaled_X
amount_initial_centers = 2
initial_centers = kmeans_plusplus_initializer(scaled_X, amount_initial_centers).initialize()
xmeans_instance = xmeans(scaled_X, initial_centers=initial_centers, )
xmeans_instance.process()
clusters = xmeans_instance.get_clusters()
clusters
len(clusters)
inversed_X = scaler.inverse_transform(scaled_X)
inversed_X
centers = xmeans_instance.get_centers()
centers
inversed_centers = scaler.inverse_transform(centers)
inversed_centers
'''
visualizer = cluster_visualizer()
visualizer.append_clusters(clusters, inversed_X, markersize = 10)
visualizer.append_cluster(inversed_centers,
None, marker='*',
markersize=20,
color='black')
visualizer.show()
'''
df_list = []
ABC_list = [chr(ord("A")+i) for i in range(26)]
for my_list, my_label in zip(clusters, ABC_list):
df_buf = df.iloc[my_list].copy()
df_buf['pred'] = my_label
df_list.append(df_buf)
DF = pd.concat(df_list)
DF.sort_index(axis='index', inplace=True)
DF
df_target = pd.DataFrame(wine_data.target, columns=['target'])
DF2 = pd.concat([DF, df_target], axis=1)
DF2.to_csv('xmeans.csv')
DF2
import seaborn as sns
y_name = 'pred'
name_list = ["alcohol", "flavanoids","color_intensity", y_name]
sns.set_context('talk')
ax = sns.pairplot(
DF2[name_list],
hue = y_name,
palette = 'gnuplot2',
kind = 'reg',
markers = '.',
diag_kind = 'kde',
diag_kws = dict(shade = True),
)
plt.savefig("xmeans_pred.png")
plt.close()
y_name = 'target'
name_list = ["alcohol", "flavanoids","color_intensity", y_name]
sns.set_context('talk')
ax = sns.pairplot(
DF2[name_list],
hue = y_name,
palette = 'gnuplot2',
kind = 'reg',
markers = '.',
diag_kind = 'kde',
diag_kws = dict(shade = True),
)
plt.savefig("xmeans_target.png")
plt.close()
以上
<広告>
リンク


